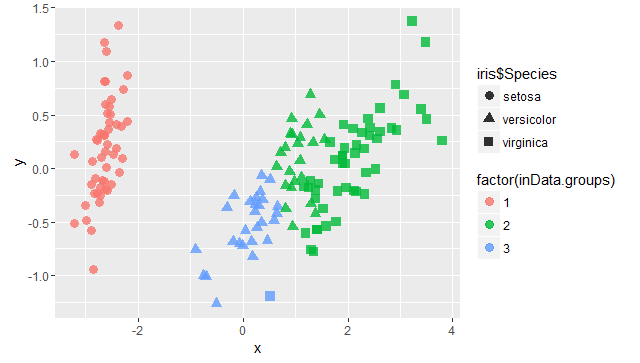
1、分层聚类
#载入R自带的测试数据 #data(iris) #attach(iris) inData = iris[,1:4] #计算距离矩阵,并绘图 inData.dist = dist(inData) #inData.dist = dist(inData,method='euclidean') heatmap(as.matrix(inData.dist), labRow = F, labCol = F) #进行分层聚类 #并绘图 #inData.hc <- hclust(inData.dist) #inData.hc <- hclust(inData.dist,method='ward') plot(inData.hc, labels = FALSE, hang = -1) #标识聚类结果,结果设为3类 rect.hclust(inData.hc, k = 3) #将Tree进行分组 inData.groups <- cutree(inData.hc, 3) #输出结果表格 table(inData.groups, Species) #进行降维处理 #绘图对比结果 #形状是正确的数据 #颜色为聚类后的数据 mds=cmdscale(inData.dist,k=2,eig=T) x = mds$points[,1] y = mds$points[,2] library(ggplot2) p=ggplot(data.frame(x,y),aes(x,y)) p+geom_point(size=3,alpha=0.8,aes(colour=factor(inData.groups),shape=iris$Species))
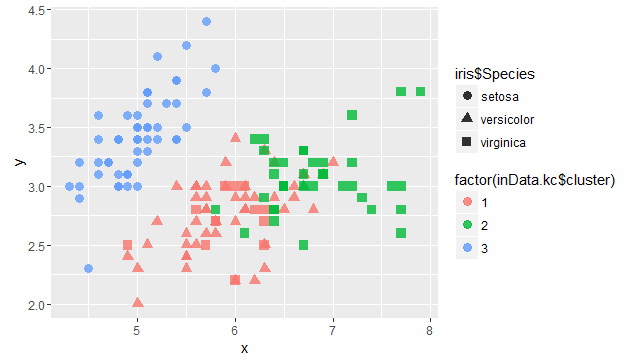
2、K值聚类
#载入R自带的测试数据
#data(iris)
#attach(iris)
inData = iris[,1:4]
#计算距离矩阵,并绘图
inData.dist = dist(inData)
#inData.dist = dist(inData,method='euclidean')
heatmap(as.matrix(inData.dist), labRow = F, labCol = F)
#进行K值聚类
inData.kc <- kmeans(inData.dist,centers=3)
#绘图对比结果
#形状是正确的数据
#颜色为聚类后的数据
library(ggplot2)
x=inData[c("Sepal.Length")]
y=inData[c("Sepal.Width")]
p=ggplot(data.frame(x,y),aes(x,y))
p+geom_point(size=3,alpha=0.8,aes(colour=factor(inData.kc$cluster),shape=iris$Species))